抗生素耐药性(Antimicrobial Resistance, AMR)是21世纪全球公共卫生面临的重大挑战之一。随着抗生素的广泛使用,耐药菌株和抗生素耐药基因(Antibiotic Resistance Genes, ARGs)在医院环境中迅速扩散,尤其是在重症监护病房(ICU)这样高风险的医疗场所。ICU医护人员因长期暴露于高浓度抗生素和耐药菌的环境中,其肠道微生物群是否受到显著影响,成为一个备受关注的研究课题。2025年发表在《Critical Care》期刊上的一篇多中心、跨区域研究通过宏基因组学技术,系统比较了ICU医护人员与健康对照组的肠道ARG丰度及微生物群组成,揭示了ICU医护人员肠道ARG显著升高的现象。
Paper Info

- 标题:Elevated antibiotic resistance gene abundance of ICU healthcare workers, a multicentre, cross-sectional study
- 译名:多中心横断面研究揭示ICU医护人员抗生素耐药基因丰度升高
- 期刊:Critical Care (IF:8.8)
- 发表时间:2025年4月30日
- 链接:https://doi.org/10.1186/s13054-025-05408-5
引言
抗生素耐药性被世界卫生组织列为全球十大公共卫生威胁之一。据《Lancet》2024年报道,1990年至2021年间,全球因AMR导致的死亡人数持续上升,预计未来几十年将进一步加剧。医院作为抗生素使用最密集的场所,是ARG传播的主要来源。ICU尤为突出,因其患者病情危重、抗生素使用频繁、耐药菌(如ESKAPE病原体:粪肠球菌、肺炎克雷伯菌等)高发,导致环境中的ARG浓度远高于外部环境。
ICU医护人员每天接触高危患者、污染表面及空气中的耐药菌,面临较高的职业暴露风险。先前的研究表明,医护人员可能通过直接接触、空气传播或交叉污染成为耐药菌的携带者。然而,传统研究多依赖细菌培养技术,样本量小,难以全面揭示ARG的传播动态。宏基因组学技术的兴起为研究ARG提供了更精确的工具,使研究人员能够深入分析医护人员肠道微生物群的耐药基因特征。
该研究提出一个核心问题:ICU医护人员的肠道ARG丰度是否高于健康人群?此外,ICU职业暴露的时长是否与ARG丰度相关?通过多中心、跨区域的样本采集和宏基因组学分析,研究旨在填补这一领域的知识空白,为医院感染防控提供科学依据。
研究设计与方法
这项前瞻性、多中心、横断面研究在中国浙江和河南的八家医疗中心开展,时间为2024年1月至2月。研究对象包括191名ICU医护人员(医生、护士、护理助理)和99名健康对照组,共290人。健康对照组来自常规体检人群,无医疗相关工作背景。研究通过粪便样本的宏基因组测序,比较两组的肠道ARG丰度、ARG多样性及微生物群组成。
- 纳入标准:年龄大于18岁,签署知情同意书。
- 排除标准:存在胃肠道疾病、恶性肿瘤、精神疾病;近6个月使用广谱抗生素(如万古霉素、头孢吡肟等);孕妇;健康对照组需无医疗相关工作经历。
研究团队收集粪便样本,提取DNA后使用Illumina NovaSeq X Plus平台进行双末端测序(2×150 bp)。通过Fastp去除低质量读数,Bowtie2过滤人类基因组序列,Kraken2进行分类学分析,RGI结合CARD数据库识别ARG。ARG丰度以每千碱基每百万映射读数(RPKM)标准化,ARG丰富度以识别的ARG总数量化。研究还通过Kraken2和RGI的读数ID匹配,追踪ARG的细菌宿主。
主要结局为比较ICU医护人员与健康对照组的ARG丰度(以RPKM为指标)。使用广义线性模型(GLM)进行分析,矫正了年龄、性别和BMI等混杂因素。次要结局为探索ICU职业暴露时长与ARG丰度的关系,使用限制性立方样条(RCS)模型。亚组分析和敏感性分析进一步验证结果的稳健性,分析工具包括R软件及其相关包(如pctax、rms)。
结果
ICU医护人员肠道ARG丰度显著升高

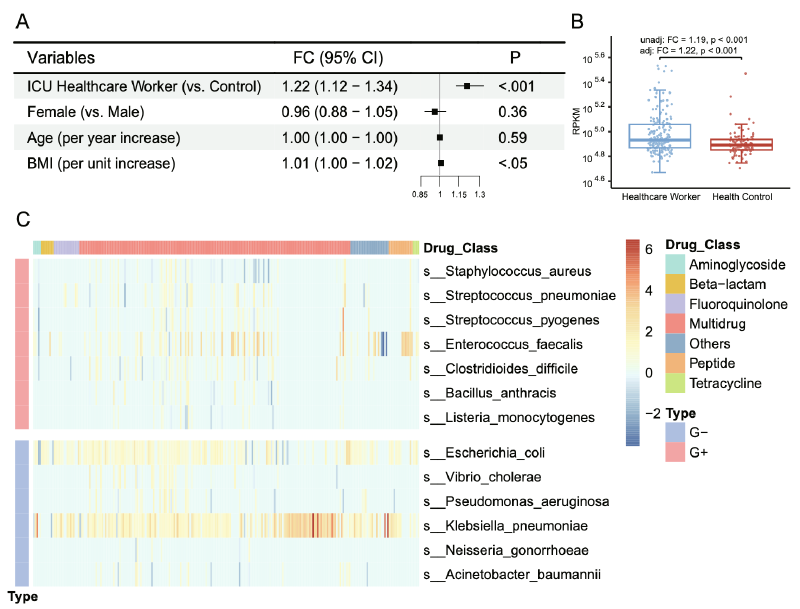
研究发现,ICU医护人员的总ARG丰度显著高于健康对照组(调整后倍数变化=1.22,95%置信区间:1.12-1.34,p<0.001)。具体而言,来源于粪肠球菌、肺炎克雷伯菌和肠杆菌属(包括大肠杆菌)的ARG丰度在医护人员中显著增加。特别值得注意的是,与喹诺酮类抗生素耐药相关的qnrE1基因(log2倍数变化=14.03,p-adj=0.02)以及与β-内酰胺类抗生素耐药相关的ACT-12(log2倍数变化=11.23,p-adj=0.03)和MIR-12(log2倍数变化=11.17,p-adj=0.04)基因在医护人员中显著上调。
ARG多样性与微生物群组成差异

ICU医护人员的ARG α多样性(丰富度和Shannon指数)显著高于对照组(p<0.001,p=0.02),β多样性也存在显著差异(Adonis R²=0.0396,p=0.001)。243个ARG在医护人员中显著上调,而仅9个ARG下调。此外,医护人员的肠道细菌组成显示出更高的α多样性(p=0.04)和β多样性(Adonis R²=0.0419,p=0.001),其中克雷伯菌属(如Klebsiella grimontii,log2倍数变化=5.93,p-adj=0.004)和乳酸乳球菌(log2倍数变化=5.78,p-adj=0.009)显著上调。
职业暴露时长与ARG丰度无关

出乎意料的是,研究未发现ICU职业暴露时长与ARG丰度之间的显著线性或非线性关系(p for overall=0.96,p for nonlinear=0.84)。这表明ARG丰度的增加可能在医护人员职业生涯的早期即已发生。
4. 亚组与敏感性分析

- 性别差异:男性医护人员的ARG丰度与职业暴露的关联更强(β=0.29,p<0.001)相比女性(β=0.13,p=0.03)。
- 地区差异:河南的医护人员ARG丰度高于浙江(倍数变化=1.20 vs. 1.12)。
- 职业差异:护理助理的ARG丰度最高(倍数变化=1.33),其次为医生(1.18)和护士(1.08)。
讨论
临床意义
- 感染防控的新视角:ICU医护人员作为潜在的ARG携带者,可能在医院内外的耐药菌传播中扮演重要角色。研究提示需加强对医护人员的感染防控措施,如优化手部卫生、加强环境消毒。
- 精准医疗的潜力:宏基因组学技术为监测ARG提供了高分辨率工具,可用于识别高风险人群和制定针对性的干预措施。
- 职业健康保障:医护人员肠道微生物群的变化可能影响其长期健康,需进一步研究这些变化的临床意义。
公共卫生意义
研究结果支持“One Health”框架,强调人类、动物和环境健康的互联性。医院作为ARG的“热点”,其防控策略应扩展到医护人员和社区。减少不必要的抗生素使用、加强耐药性监测是应对AMR的关键。
局限性
- 地域局限:研究仅限于中国人群,结论的普遍性需进一步验证。
- 因果关系:作为观察性研究,无法确定职业暴露与ARG升高之间的因果关系。
- 未测混杂因素:如旅行史、住院史等可能影响结果。
- 临床相关性:ARG的升高是否导致实际感染风险尚不明确。
未来研究方向
- 纵向研究:通过长期随访,探索ARG丰度变化的动态和影响因素。
- 全球合作:开展跨国研究,验证不同地区医护人员的ARG特征。
- 干预措施:测试感染防控策略(如益生菌干预、环境净化)对ARG丰度的影响。
- 健康影响:研究ARG升高对医护人员健康的潜在影响,如免疫功能或代谢疾病风险。
小结
这项多中心研究揭示了ICU医护人员肠道ARG丰度和微生物群多样性的显著升高,提示职业暴露可能是重要驱动因素。尽管未发现暴露时长与ARG丰度的直接关联,但研究强调了医院环境在耐药基因传播中的关键作用。宏基因组学技术的应用为理解AMR动态提供了新视角,未来需进一步探索其临床和公共卫生意义,以优化感染防控策略,保护医护人员和患者的健康。
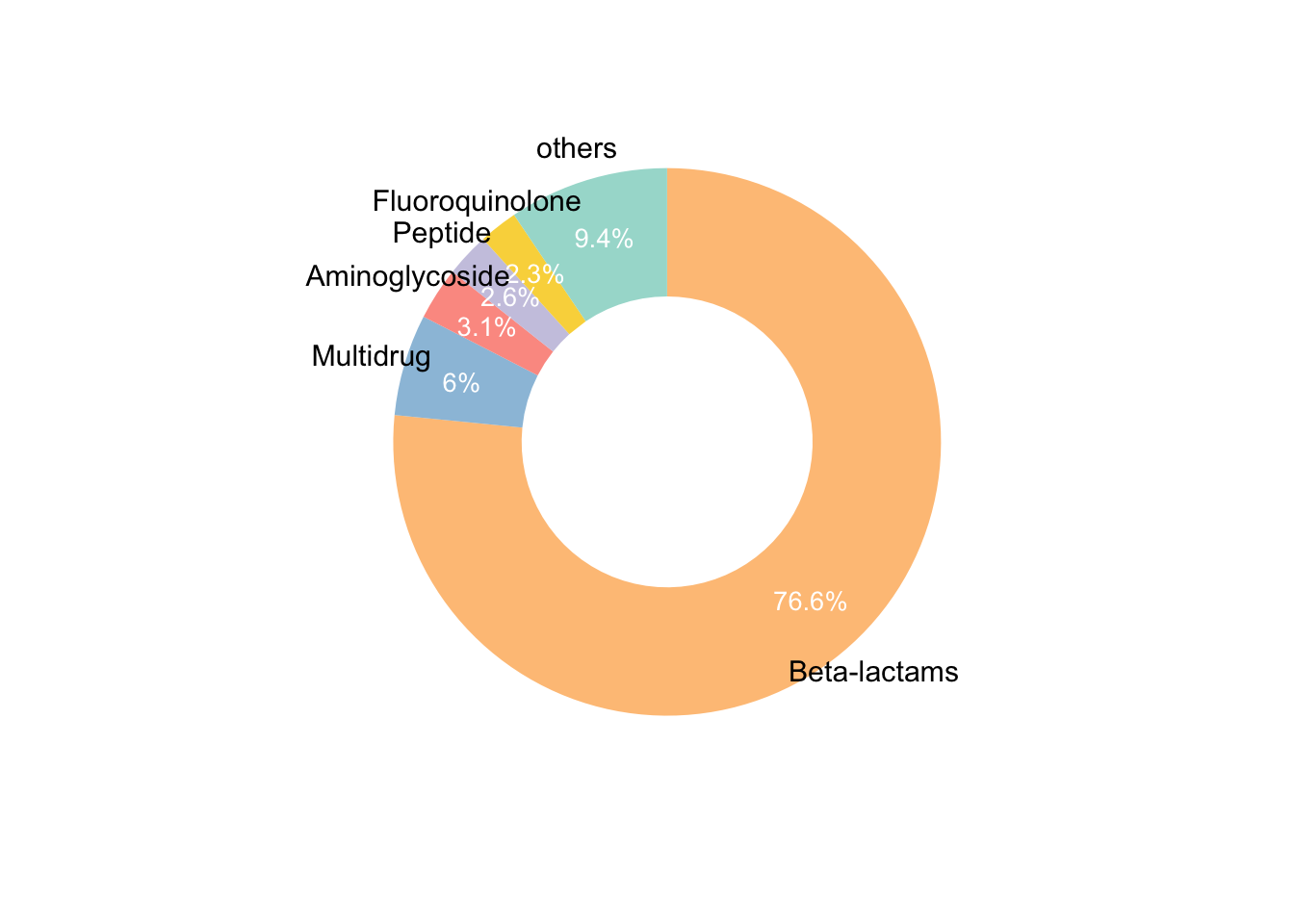
CARD数据库中药物类型整理
为了方便分析,我整合了CARD数据库的描述信息到ReporterScore包中,可以通过以下代码调取:
|
|
|
|
注意CARD数据库中Drug Class,一个ARO可以对应很多种药物类型,不是很方便整合,所以我根据《Assessment of global health risk of antibiotic resistance genes》中的规则新增了一列“Drug_Class”。
ARGs were manually reclassified based on the drugs to which they confer resistance. ARGs referring to penam, cephalosporin, carbapenem, cephamycin, penem and monobactam were grouped into the beta-lactam class. ARGs referring to macrolides, lincosamides and streptogramins were grouped into the MLS class. ARGs referring to more than one drug class were grouped into the multidrug class.
|
|