Introduction
MetaNet是一个用于组学网络分析的R包,提供了多种功能,包括网络构建、可视化、比较和稳定性分析等。
之前发布的推文中,有多位同学提到如何进行网络的比较,我也根据他们的一些建议改进了MetaNet的一些函数。
本文将介绍如何使用MetaNet进行网络比较,并展示一些示例代码和结果。
可以从 CRAN 安装稳定版:install.packages("MetaNet"),本文请使用最新的开发版本。
最新的开发版本可以在 https://github.com/Asa12138/MetaNet 中找到:
1
remotes :: install_github ( "Asa12138/MetaNet" , dependencies = T )
Copy 网络间运算
多个网络之间的比较和运算操作对组学数据分析很重要,例如,比较不同组别网络之间的差异部分,或者是探究动态网络变化中的核心稳定子网络。为了方便比较,MetaNet提供了c_net_union、c_net_intersect、c_net_difference等函数来计算网络的并集、交集和差集。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
library ( MetaNet )
library ( igraph )
set.seed ( 123 )
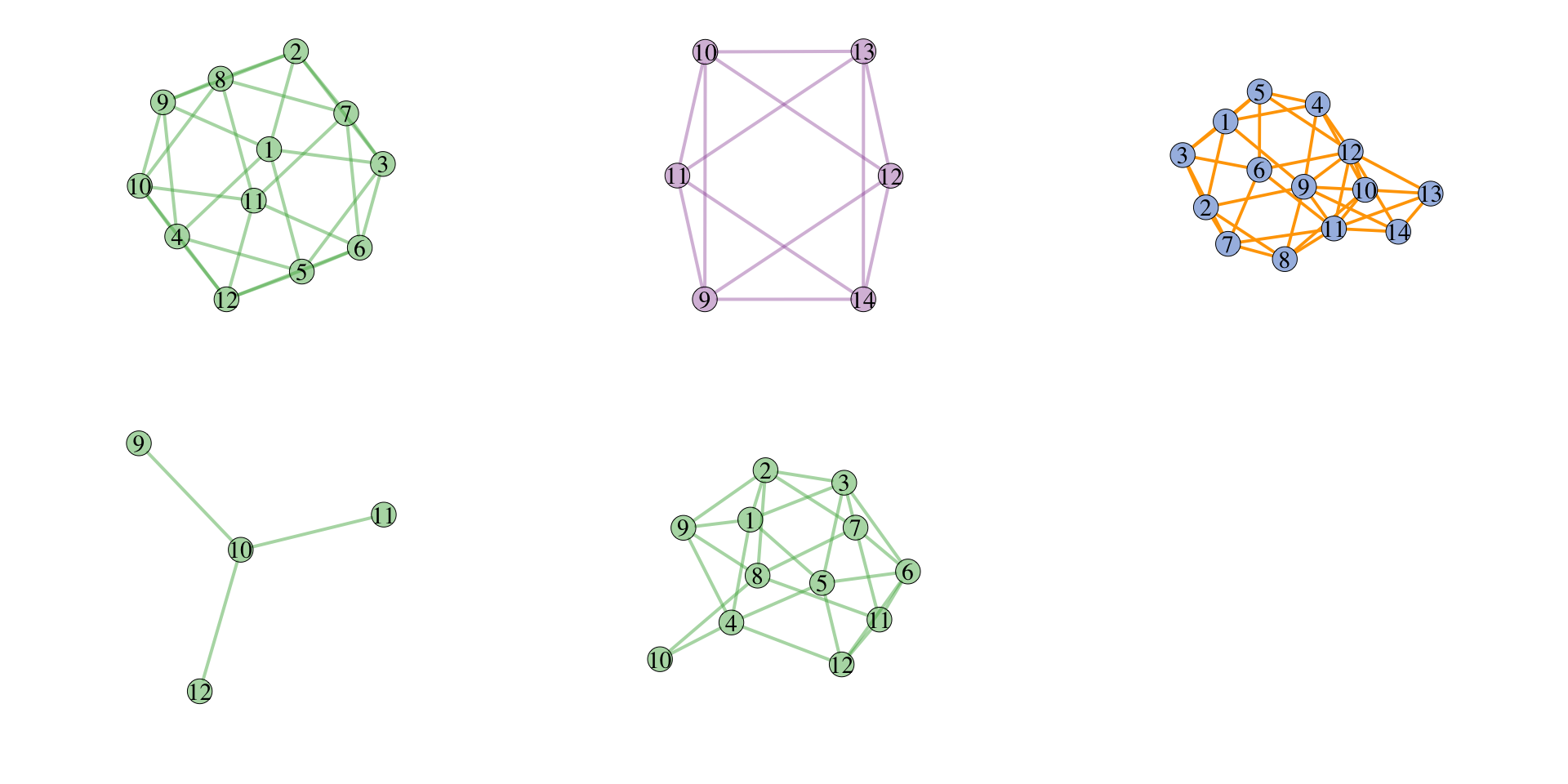
g1 <- make_graph ( "Icosahedron" )
V ( g1 ) $ color <- "#4DAF4A77"
E ( g1 ) $ color <- "#4DAF4A77"
g1 = as.metanet ( g1 )
g2 <- make_graph ( "Octahedron" )
V ( g2 ) $ name = as.character ( 9 : 14 )
V ( g2 ) $ color <- "#984EA366" # 紫色
E ( g2 ) $ color <- "#984EA366"
g2 = as.metanet ( g2 )
# 执行操作
g_union <- c_net_union ( g1 , g2 )
E ( g_union ) $ color <- "orange"
g_inter <- c_net_intersect ( g1 , g2 )
g_diff <- c_net_difference ( g1 , g2 )
par_ls = list ( main = "" , legend = F , vertex_size_range = c ( 20 , 20 ))
par ( mfrow = c ( 2 , 3 ))
c_net_plot ( g1 , params_list = par_ls )
c_net_plot ( g2 , params_list = par_ls )
c_net_plot ( g_union , params_list = par_ls )
c_net_plot ( g_inter , params_list = par_ls )
c_net_plot ( g_diff , params_list = par_ls )
Copy c_net_compare
基于上述的网络运算,MetaNet提供了c_net_compare函数来比较两个网络的差异部分。该函数可以计算两个网络之间的并集、交集、网络拓扑指标以及计算的网络相似性,并返回一个包含这些信息的列表。
1
2
3
4
5
6
7
8
set.seed ( 12 )
co_net_p1 = c_net_filter ( co_net , name %in% sample ( V ( co_net ) $ name , 300 ))
co_net_p2 = c_net_filter ( co_net , name %in% sample ( V ( co_net ) $ name , 300 ))
c_net_compare ( co_net_p1 , co_net_p2 ) -> c_net_comp
# 展示网络拓扑指标
c_net_comp $ net_par_df
Copy
1
2
3
4
5
6
7
8
9
10
11
12
13
## g1 g2 g_union g_inter
## Node_number 300.000000000 300.000000000 392.0000000 208.000000000
## Edge_number 334.000000000 321.000000000 499.0000000 156.000000000
## Edge_density 0.007447046 0.007157191 0.0065113 0.007246377
## Negative_percentage 0.443113772 0.389408100 0.4128257 0.429487179
## Average_path_length 6.801472290 6.964863184 7.3484195 5.549893086
## Global_efficiency 0.088800074 0.067651256 0.1044344 0.033600618
## Average_degree 2.226666667 2.140000000 2.5459184 1.500000000
## Average_weighted_degree 0.787061535 0.755481803 0.8992606 0.530061699
## Diameter 18.000000000 18.000000000 19.0000000 13.000000000
## Clustering_coefficient 0.330228620 0.311132255 0.2922078 0.337595908
## Centralized_betweenness 0.127059002 0.074659531 0.1184792 0.049258854
## Natural_connectivity 3.441388267 2.895183227 4.3359822 1.643829036
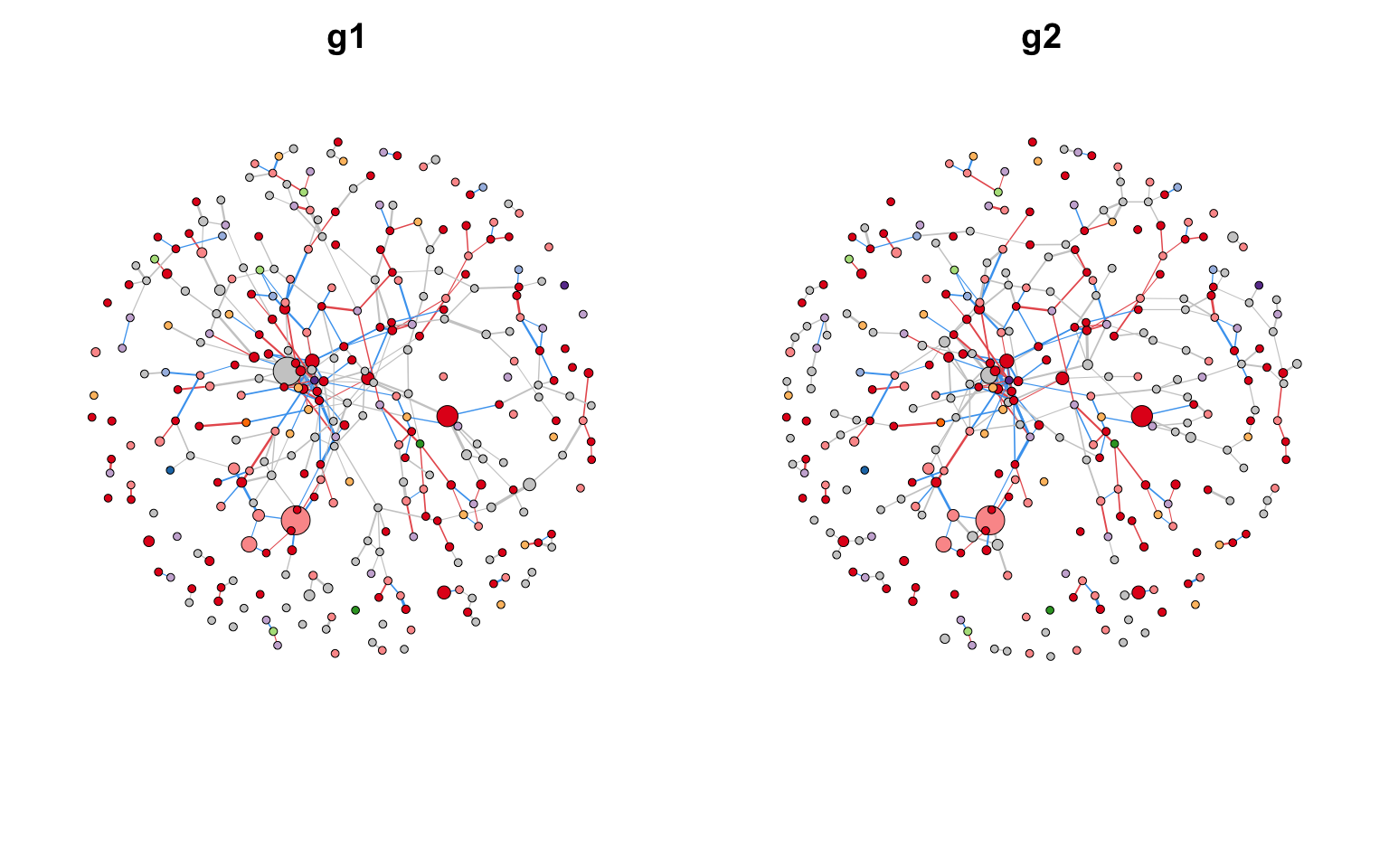
Copy 直接plot一下,结果图会将两个网络中不同的节点和边变成灰色,相当于共有节点和边高亮出来,帮助我们更好地看见网络的共有或差异部分。
网络相似性计算了三种,一个是基于共有节点的jaccard相似性,一个是基于共有边的jaccard相似性,还有一个是基于网络邻接矩阵的相似性。
1
c_net_comp $ net_similarity
Copy
1
2
## node_jaccard edge_jaccard adjacency_similarity
## 0.5306122 0.3126253 0.9331847
Copy 邻接矩阵相似性计算的实现代码如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
adjacency_similarity <- function ( g1 , g2 , method = "frobenius" ) {
if ( ! is_metanet ( g1 )) g1 <- as.metanet ( g1 )
if ( ! is_metanet ( g2 )) g2 <- as.metanet ( g2 )
# 获取邻接矩阵
adj1 <- as.matrix ( igraph :: as_adjacency_matrix ( g1 ))
adj2 <- as.matrix ( igraph :: as_adjacency_matrix ( g2 ))
# 统一节点集合
all_nodes <- union ( rownames ( adj1 ), rownames ( adj2 ))
# 初始化全零矩阵
adj1_fixed <- matrix ( 0 , nrow = length ( all_nodes ), ncol = length ( all_nodes ),
dimnames = list ( all_nodes , all_nodes ))
adj2_fixed <- matrix ( 0 , nrow = length ( all_nodes ), ncol = length ( all_nodes ),
dimnames = list ( all_nodes , all_nodes ))
# 填充已知边
adj1_fixed [rownames ( adj1 ), colnames ( adj1 ) ] <- adj1
adj2_fixed [rownames ( adj2 ), colnames ( adj2 ) ] <- adj2
# 计算相似性
if ( method == "frobenius" ) {
diff_norm <- norm ( adj1_fixed - adj2_fixed , "F" )
max_norm <- sqrt ( nrow ( adj1_fixed ) * ncol ( adj2_fixed ))
similarity <- 1 - diff_norm / max_norm
} else if ( method == "cosine" ) {
similarity <- sum ( adj1_fixed * adj2_fixed ) /
( norm ( adj1_fixed , "F" ) * norm ( adj2_fixed , "F" ))
} else {
stop ( "Method must be 'frobenius' or 'cosine'." )
}
return ( similarity )
}
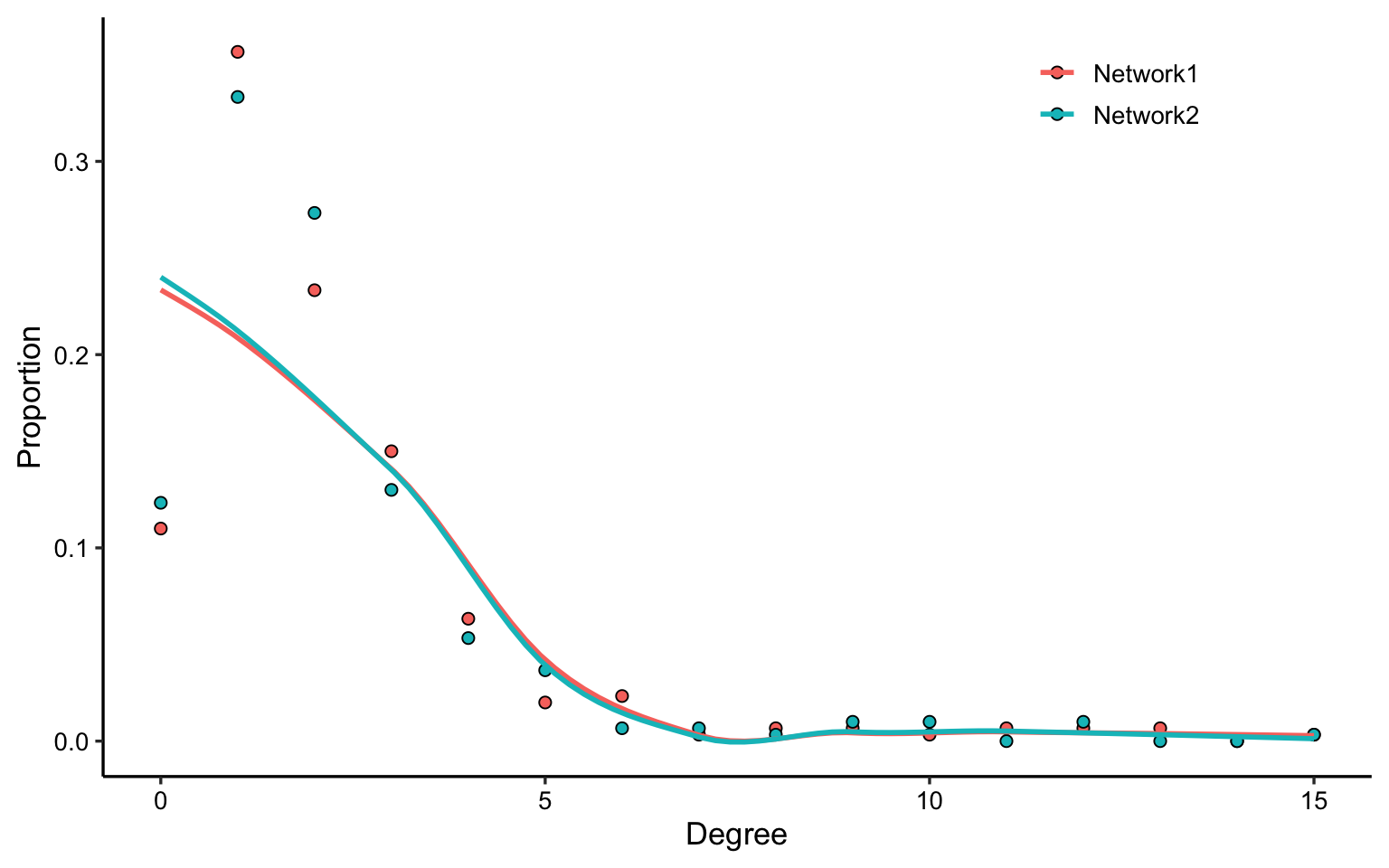
Copy 我们还可以使用plot_net_degree函数来绘制网络的度分布图,帮助更好地理解网络的结构特征,下面这两个随机取出来的子网络的度分布非常类似。
1
plot_net_degree ( list ( co_net_p1 , co_net_p2 ))
Copy c_net_highlight
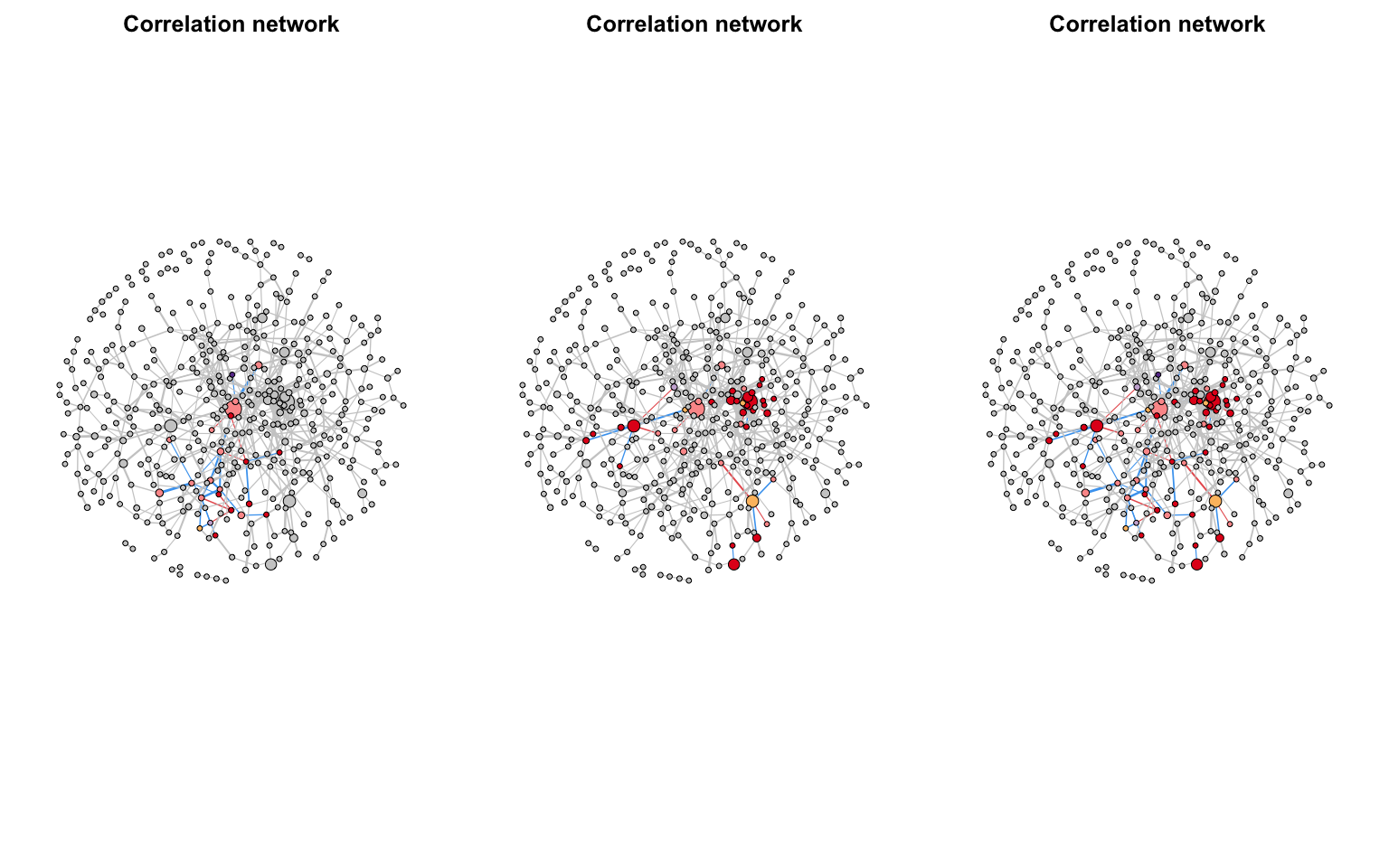
我们也可以自行调用以下这些函数:
c_net_neighbors函数可以获取指定节点的邻居节点,
c_net_highlight函数可以高亮显示网络中指定的节点和边,方便用户进行网络比较和分析。
plot_multi_nets函数可以将多个网络图并排显示,便于展示。
1
2
3
4
5
6
7
8
9
10
11
nodes <- c ( "s__Kribbella_catacumbae" , "s__Verrucosispora_andamanensis" )
nodes <- V ( c_net_neighbors ( co_net , nodes , order = 2 )) $ name
g_hl <- c_net_highlight ( co_net , nodes = nodes )
get_e ( co_net ) %>% head ( 40 ) -> hl_edges
g_hl2 <- c_net_highlight ( co_net , edges = hl_edges[ , 2 : 3 ] )
g_hl3 <- c_net_highlight ( co_net , nodes = nodes , edges = hl_edges[ , 2 : 3 ] )
plot_multi_nets (
list ( g_hl , g_hl2 , g_hl3 ), nrow = 1 , multi_params_list = list ( list ( legend = F ))
)
Copy